医学部医学科基礎医学系分子生命科学領域の中川草准教授(総合医学研究所/マイクロ・ナノ研究開発センター)が大阪大学微生物病研究所等との共同研究で、新型コロナウイルスのnsp14遺伝子の変異がゲノム多様性を向上させる可能性があることを解明。その成果をまとめた論文が2月15日に、科学雑誌『iScience』オンライン版に掲載されました。
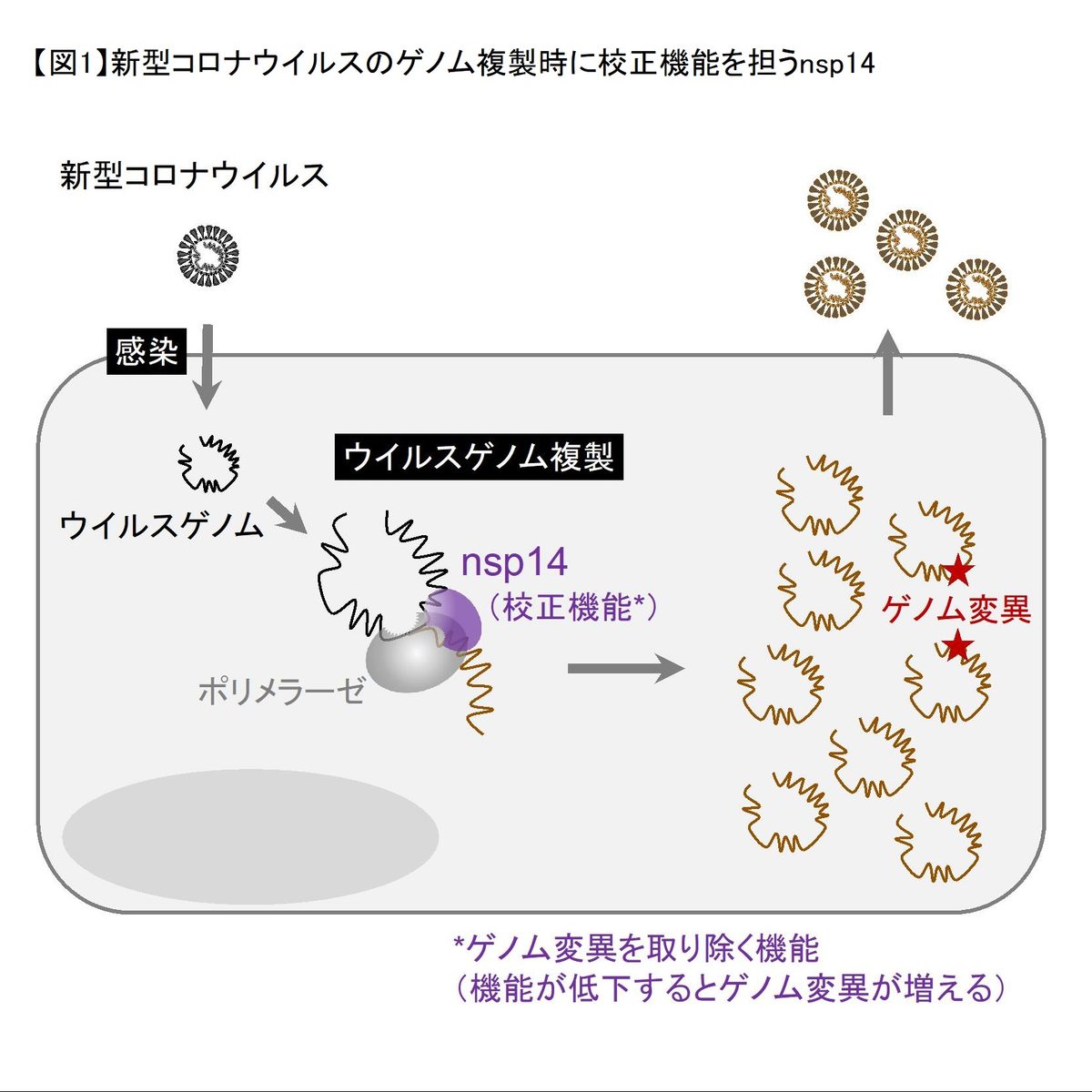
風邪などの原因となるコロナウイルスは、ゲノムを複製する際に生じたエラーを校正する機能を持つ遺伝子(nsp14)を有しているため、ウイルス増殖時に変異が起きにくいと考えられています。しかし、新型コロナウイルスについては多様なゲノム変異が確認されたため、研究グループでは校正機能が低下したnsp14を持つ新型コロナウイルスの存在を予測。まず、62種類のコロナウイルスを対象にnsp14のアミノ酸配列の進化を分子レベルで解析し、コロナウイルスでは進化の過程において同配列が比較的よく保存され、ゲノム変異が起こりにくい(種の保存に有害な遺伝子変異があった場合には集団から除去する力が強い)ことを明らかにしました。
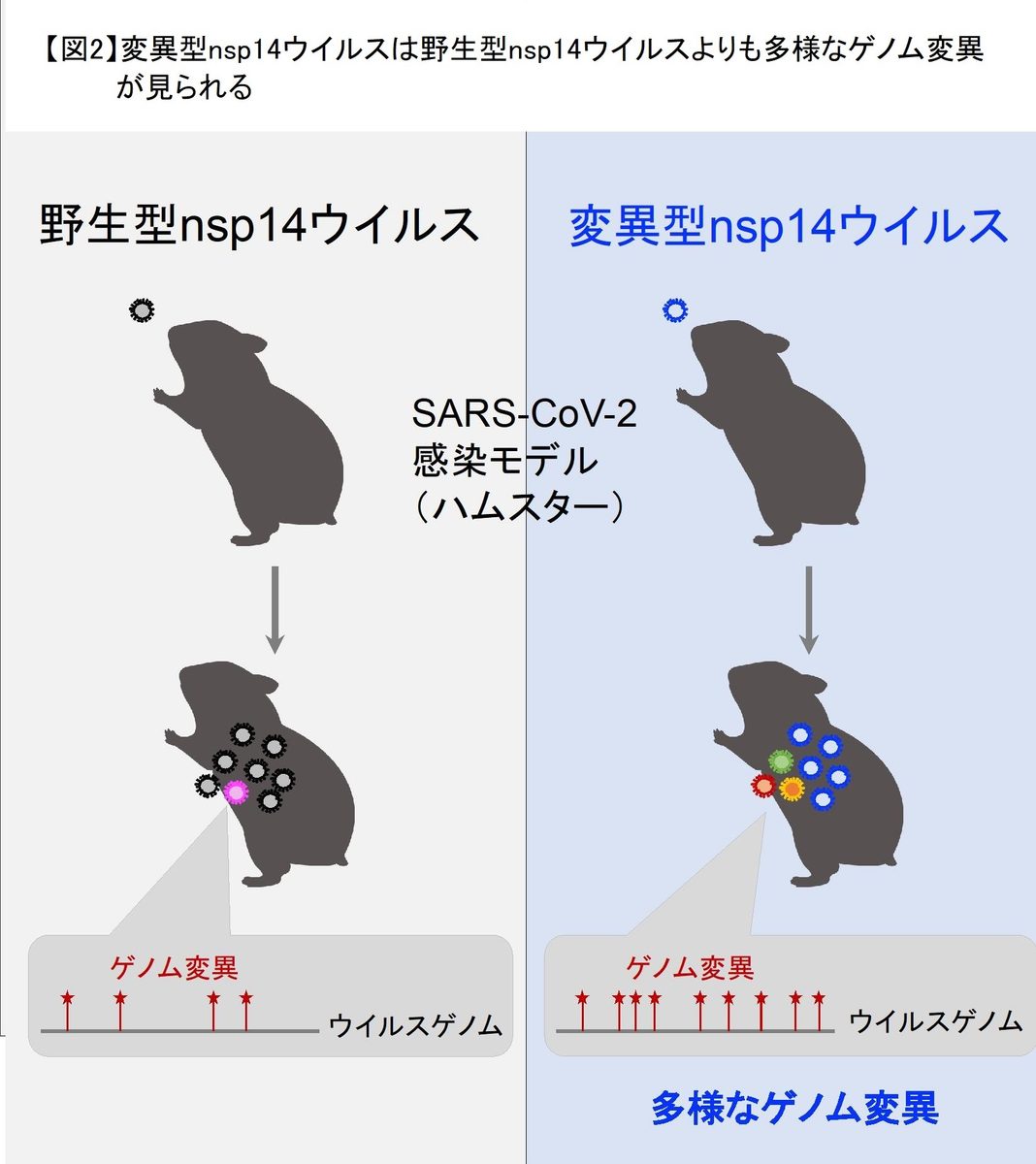
次に、新型コロナウイルスの大規模データ解析により、nsp14-P203Lというアミノ酸置換を持つウイルス株は、ゲノム進化速度が大きい傾向があることを発見。この結果を確認するため、nsp14-P203Lを持つウイルス(変異型nsp14ウイルス)と野生型nsp14を持つウイルス(野生型nsp14ウイルス)を作出してハムスターに感染させ、肺で増殖したウイルスのゲノム変異を解析したところ、変異型nsp14ウイルスは野生型よりも多様なゲノム変異が見られることを突き止めました。同時に、nsp14-P203Lのようにゲノム校正機能が低下した変異型nsp14を持つウイルスの出現は稀であることも見いだしました。
中川准教授は、「本研究により、コロナウイルスのnsp14がゲノムの維持に重要な役割を持つと同時に、nsp14の機能維持がコロナウイルスの適応と進化に欠かせないことが明らかになりました。この成果は、ゲノムデータ解析とウイルス実験の組み合わせによってウイルスの進化の一端をひもといたものであり、この手法は遺伝子変異によるウイルスの性状変化を明らかにするための有効な手段になると期待されます」と話しています。
※『iScience』に掲載された論文は下記URLからご覧いただけます。
https://www.cell.com/iscience/fulltext/S2589-0042(23)00287-0